
Pequenos fragmentos de DNA podem contar histórias importantes, especialmente quando são originados de células cancerígenas. Mutações transportadas pela corrente sanguínea em pedaços dispersos de DNA podem denunciar a presença de um tumor nascente, que está crescendo além dos limites convencionais de detecção. Outras mutações encontradas no DNA livre de células podem indicar um tumor que está evoluindo para resistência terapêutica. Naturalmente, o DNA tumoral circulante (ctDNA) e as histórias que ele guarda se tornaram uma fonte promissora de biomarcadores clínicos, com aplicações emergentes na detecção precoce de câncer, orientação do tratamento e testes de doença residual mínima.1-3
No entanto, a natureza elusiva do ctDNA torna seu acesso extremamente difícil. O plasma sanguíneo, a urina e outros fluidos corporais usados para biópsias líquidas estão saturados com DNA livre de células (cfDNA) que foi liberado de tecidos saudáveis por todo o corpo. Dentro desse meio genético, apenas cerca de 0,01% a 10% dos fragmentos totais de cfDNA são provavelmente reconhecidos como derivados de tumor. Além disso, não está sempre claro como o ctDNA será: mutações associadas ao tumor podem ocorrer em milhares de locais potenciais.
O valor e o potencial das biópsias líquidas são temas de pesquisa em andamento. Para detectar ctDNA de forma confiável, os pesquisadores precisam, portanto, ter a capacidade de limitar o escopo do sequenciamento a fragmentos raros derivados de tumor—para não desperdiçar uma quantidade substancial de recursos de sequenciamento em fragmentos de DNA livre de células irrelevantes—enquanto também lançam uma rede ampla para garantir que todas as mutações potenciais sejam contabilizadas. Felizmente, os recentes avanços na tecnologia de sequenciamento de próxima geração ajudaram a melhorar a sensibilidade e a especificidade da detecção de ctDNA, permitindo a otimização tanto da preparação da biblioteca quanto do enriquecimento alvo. A Twist Bioscience tem um kit de preparação de biblioteca (o Twist cfDNA Library Preparation Kit) que é projetado especificamente para apoiar a pesquisa de ctDNA.
O Valor do ctDNA
Fragmentos circulantes de DNA tumoral são cada vez mais vistos como fontes valiosas de informação clínica, com aplicações potenciais em triagem precoce de câncer, testes de doença residual mínima (MRD), perfilagem tumoral e tomada de decisões terapêuticas.1-3
Apenas pela sua presença, o ctDNA pode ser informativo, indicando a possível existência de uma célula maligna. Teoricamente, o ctDNA pode ser liberado de uma única célula cancerosa, abrindo a possibilidade de detectar crescimentos malignos em seus estágios mais iniciais. Embora ainda existam desafios significativos para o desenvolvimento de um teste tão sensível, várias aplicações clínicas do ctDNA são baseadas nesse princípio. Por exemplo, o ctDNA está se mostrando um biomarcador altamente sensível para testes de doença residual mínima.1,6 Vários estudos mostraram que o ctDNA pode prever a recorrência do tumor, permitindo a detecção de tumores recorrentes meses antes de se tornarem detectáveis por meios convencionais (em alguns casos, o ctDNA identificou tumores que nunca atingiram os limiares de detecção clínica).1,2,7,8 O sucesso desses testes levanta a possibilidade real de usar rotineiramente o ctDNA para MRD e triagem precoce de câncer.
Igualmente importante é a ausência de ctDNA. Após tratamentos curativos, muitos pacientes recebem proativamente terapia adjuvante na tentativa de prevenir a recorrência do tumor. No entanto, essas terapias são severas e podem causar danos. Foi descoberto que um teste de MRD negativo está correlacionado com uma menor probabilidade de recorrência do tumor e menor necessidade de terapia adjuvante. Portanto, um resultado verdadeiro-negativo pode ser usado para poupar os pacientes de danos desnecessários.9,10
Além disso, o ctDNA traz naturalmente informações sobre sua origem maligna na forma de mutações e assinaturas epigenéticas. As primeiras podem ser usadas para construir perfis mutacionais que expõem as vulnerabilidades terapêuticas do tumor (ou a falta delas).4,11 Com as segundas, padrões de metilação podem ser usados para determinar de qual tecido o ctDNA foi liberado. Essas informações podem ser úteis na detecção de tumores metastáticos ou nascente.
Para desenvolver e refinar ainda mais essas aplicações potenciais, são necessárias pesquisas significativas, bem como avanços nas ferramentas usadas para realizá-las.
Preparando-se para o Impacto
A raridade do ctDNA apresenta desafios substanciais para sua detecção.5 Painéis de captura de alvo de alta qualidade podem ser usados para enriquecer efetivamente amostras para fragmentos de ctDNA, permitindo sequenciamento mais profundo (e, portanto, com maior confiança). No entanto, mesmo o melhor fluxo de trabalho de enriquecimento de alvo pode ser prejudicado por uma preparação de biblioteca deficiente.
A preparação da biblioteca é a etapa crítica em qualquer fluxo de trabalho de sequenciamento onde o material genético alvo (neste caso, ctDNA) é tornado adequado para sequenciamento de próxima geração (NGS). Isso envolve a reparação de extremidades de fita simples em fragmentos de DNA, a ligação de identificadores moleculares únicos e adaptadores de NGS a cada extremidade, e a subsequente amplificação por PCR. Ao longo desse processo, existem várias oportunidades para que erros ocorram, o que pode diminuir substancialmente a sensibilidade e a especificidade do teste. Duas principais fontes de erro são:
- Conversão Deficiente: Reparos imperfeitos nas extremidades e ineficiências na ligação reduzem o número de fragmentos que são devidamente convertidos para um formato pronto para sequenciamento. Esta é a fonte mais significativa de erro e pode reduzir muito a sensibilidade do teste.5 A conversão falhada pode potencialmente eliminar os poucos fragmentos de ctDNA que estão presentes e levar a um resultado falso-negativo.
- Erros de PCR: As polimerases cometem erros aproximadamente duas vezes a cada cem mil pares de base amplificados.12 Quando esses erros ocorrem no início do processo de PCR, a amplificação subsequente do erro pode fazê-lo parecer legítimo, levando a um resultado falso-positivo.
A Twist desenvolveu um kit de preparação de biblioteca (o Twist cfDNA Library Preparation Kit) que é projetado para minimizar erros, maximizar a taxa de conversão e permitir uma análise confiante de ctDNA. Isso é alcançado através de várias otimizações, incluindo reações de reparo de extremidade e ligação de adaptador cuidadosamente ajustadas, uso de uma polimerase de alta fidelidade e compatibilidade do fluxo de trabalho com sequenciamento duplex.
🧬 O que é o sequenciamento duplex?
O sequenciamento duplex é uma maneira elegante de minimizar falsos positivos provenientes de erros de polimerase e sequenciamento. O objetivo principal do sequenciamento duplex é utilizar leituras de ambas as fitas de uma sequência de DNA complementar para validar os resultados. Durante a análise bioinformática, leituras de ambas as fitas da molécula de origem são usadas para corrigir erros e eliminar falsos positivos. Quando há discordância entre as duas fitas complementares, pode-se inferir que ocorreu um erro, e a mutação observada é artificial.
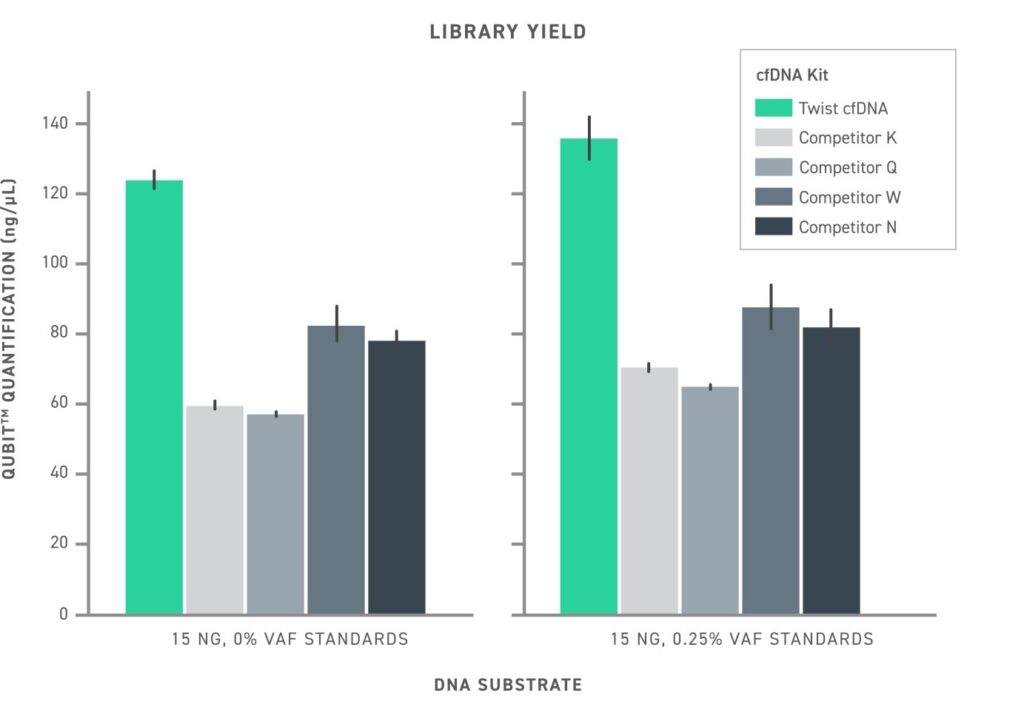
Em comparação com outros kits de preparação de biblioteca de NGS recomendados para pesquisa de cfDNA (Figura 1), a abordagem da Twist leva a uma maior taxa de conversão da biblioteca (finalmente medida como rendimento da biblioteca) e a uma complexidade geral aumentada da biblioteca. Ao estudar amostras de biópsia líquida, onde o número de fragmentos de ctDNA pode ser menor que 10, ter uma alta taxa de conversão é crucial para a sensibilidade do teste.
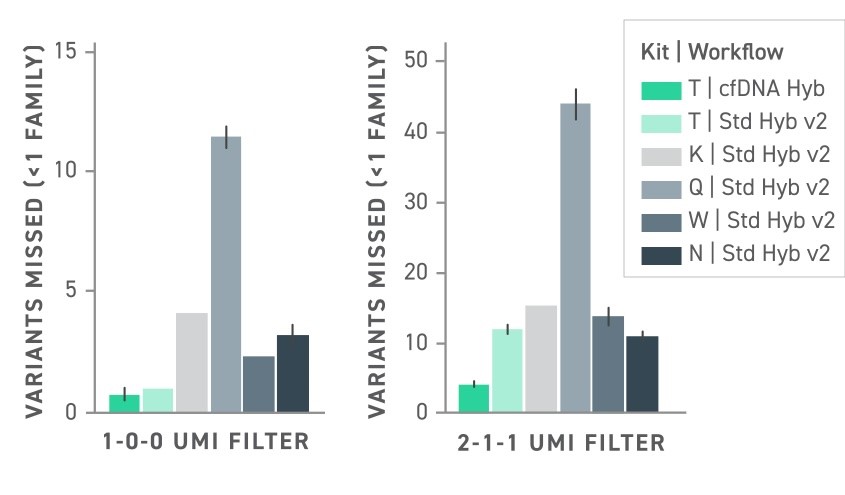
Isso é enfatizado ao tentar detectar variantes de baixa frequência em DNA livre de células (Figura 2). Seja utilizando sequenciamento duplex ou não, o Kit de Preparação de Biblioteca Twist cfDNA supera os concorrentes por uma ampla margem, demonstrando um número muito reduzido de variantes não detectadas.
O fluxo de trabalho otimizado de preparação de biblioteca da Twist (particularmente quando combinado com um painel de captura por hibridização da Twist) capacita os pesquisadores a superar alguns dos principais desafios que têm dificultado a pesquisa de ctDNA. Dessa forma, a Twist está desbloqueando o campo, permitindo o desenvolvimento de testes de ctDNA cada vez mais sensíveis para uma gama crescente de aplicações.
Acesso o artigo original e em Inglês:
https://www.twistbioscience.com/blog/science/cfDNA-library-preparation-kit
Referências
- Bronkhorst, Abel Jacobus, et al. “The Emerging Role of Cell-Free DNA as a Molecular Marker for Cancer Management.” Biomolecular Detection and Quantification, vol. 17, Mar. 2019, p. 100087, https://doi.org/10.1016/j.bdq.2019.100087.
- Abbosh, Christopher, et al. “Phylogenetic CtDNA Analysis Depicts Early-Stage Lung Cancer Evolution.” Nature, vol. 545, no. 7655, 26 Apr. 2017, pp. 446–451, www.nature.com/articles/nature22364, https://doi.org/10.1038/nature22364.
- Peng, Yan, et al. “Circulating Tumor DNA and Minimal Residual Disease (MRD) in Solid Tumors: Current Horizons and Future Perspectives.” Frontiers in Oncology, vol. 11, 18 Nov. 2021, www.ncbi.nlm.nih.gov/pmc/articles/PMC8637327/, https://doi.org/10.3389/fonc.2021.763790.
- Wan, Jonathan C. M., et al. “Liquid Biopsies Come of Age: Towards Implementation of Circulating Tumour DNA.” Nature Reviews. Cancer, vol. 17, no. 4, 1 Apr. 2017, pp. 223–238, https://doi.org/10.1038/nrc.2017.7.
- Song, Ping, et al. “Limitations and Opportunities of Technologies for the Analysis of Cell-Free DNA in Cancer Diagnostics.” Nature Biomedical Engineering, vol. 6, no. 3, 1 Mar. 2022, pp. 232–245, pubmed.ncbi.nlm.nih.gov/35102279/, https://doi.org/10.1038/s41551-021-00837-3.
- Faulkner, Lucy G., et al. “The Utility of CtDNA in Detecting Minimal Residual Disease Following Curative Surgery in Colorectal Cancer: A Systematic Review and Meta-Analysis.” British Journal of Cancer, vol. 128, no. 2, 8 Nov. 2022, pp. 297–309, https://doi.org/10.1038/s41416-022-02017-9.
- Zoe June Assaf, et al. “A Longitudinal Circulating Tumor DNA-Based Model Associated with Survival in Metastatic Non-Small-Cell Lung Cancer.” Nature Medicine, vol. 29, no. 4, 16 Mar. 2023, pp. 859–868, www.ncbi.nlm.nih.gov/pmc/articles/PMC10115641/, https://doi.org/10.1038/s41591-023-02226-6.
- Chaudhuri, Aadel A, et al. “Early Detection of Molecular Residual Disease in Localized Lung Cancer by Circulating Tumor DNA Profiling.” Cancer Discovery, vol. 7, no. 12, 2017, pp. 1394–1403, www.ncbi.nlm.nih.gov/pubmed/28899864, https://doi.org/10.1158/2159-8290.CD-17-0716.
- Sanz-Garcia, Enrique, et al. “Monitoring and Adapting Cancer Treatment Using Circulating Tumor DNA Kinetics: Current Research, Opportunities, and Challenges.” Science Advances, vol. 8, no. 4, 28 Jan. 2022, https://doi.org/10.1126/sciadv.abi8618.
- Tie, Jeanne, et al. “Circulating Tumor DNA Analysis Guiding Adjuvant Therapy in Stage II Colon Cancer.” New England Journal of Medicine, vol. 386, no. 24, 16 June 2022, pp. 2261–2272, https://doi.org/10.1056/nejmoa2200075.
- Thress, Kenneth S, et al. “Acquired EGFR C797S Mutation Mediates Resistance to AZD9291 in Non–Small Cell Lung Cancer Harboring EGFR T790M.” Nature Medicine, vol. 21, no. 6, 4 May 2015, pp. 560–562, www.ncbi.nlm.nih.gov/pmc/articles/PMC4771182/, https://doi.org/10.1038/nm.3854.
- McInerney, Peter, et al. “Error Rate Comparison during Polymerase Chain Reaction by DNA Polymerase.” Molecular Biology International, vol. 2014, 2014, pp. 1–8, https://doi.org/10.1155/2014/287430.
